Nella lotta contro le malattie infettive, la resistenza agli antibiotici rimane una delle minacce più urgenti per la salute globale. Un recente studio pubblicato su Nature Communications ha evidenziato un fattore cruciale nella comparsa della multi-resistenza nei batteri: l’ipermutazione causata dalla carenza nella riparazione dei mismatch di DNA (Mismatch Repair, MMR). La ricerca, focalizzata su Pseudomonas aeruginosa, sottolinea il ruolo dell’analisi delle firme mutazionali nella previsione dell’ipermutazione batterica e, di conseguenza, nella prevenzione della multiresistenza. Questa scoperta apre nuove prospettive per la medicina di precisione, offrendo uno strumento diagnostico innovativo per guidare terapie antibiotiche mirate.
Comprendere l’ipermutazione batterica
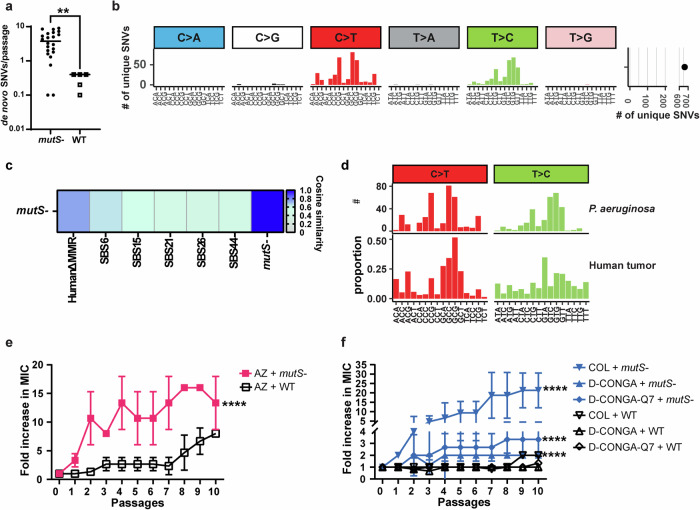
Batteri come Pseudomonas aeruginosa possono diventare ipermutatori quando perdono il loro sistema MMR. Questo meccanismo risulta essenziale per correggere gli errori di replicazione e la sua carenza porta a un aumento del tasso di mutazione. Questo fenomeno accelera la capacità del batterio di sviluppare resistenza agli antibiotici, rendendo le infezioni più difficili da trattare.
Gli ipermutatori sono spesso presenti in contesti clinici, in particolare nelle infezioni respiratorie croniche. Fino al 60% degli isolati di P. aeruginosa da pazienti con fibrosi cistica presentano una carenza nella MMR. Sebbene questa carenza sia associata alla rapida resistenza a singoli antibiotici, il suo ruolo nell’acquisizione della multi-resistenza è sempre rimasto fino ad ora poco chiaro.
Lo studio: come la carenza di MMR alimenta la resistenza
Gli scienziati hanno cercato di determinare in che modo la carenza nell’MMR contribuisca alla multiresistenza in P. aeruginosa. Utilizzando il metodo dell’evoluzione adattativa in vitro, i ricercatori hanno esposto dei ceppi batterici privi della MMR ad antibiotici di prima linea e di ultima istanza. Questo approccio è stato poi associato al sequenziamento dell’intero genoma (Whole Genome Sequencing, WGS) per analizzare la firma mutazionale associata alla carenza di MMR e tracciare lo sviluppo della resistenza.
I risultati hanno rivelato che P. aeruginosa ipermutato acquisisce la multiresistenza indipendentemente dalla classe di antibiotico. Invece di sviluppare resistenza attraverso meccanismi specifici del farmaco, i batteri si affidano a dei percorsi di resistenza comuni tra diversi farmaci. Ciò suggerisce che la monoterapia tradizionale, in cui viene utilizzato un solo antibiotico alla volta, potrebbe involontariamente accelerare la multiresistenza nei ceppi ipermutatori.
Il ruolo dell’analisi delle firme mutazionali
Uno degli aspetti chiave dello studio è il potenziale dell’analisi delle firme mutazionali come strumento predittivo. Esaminando i modelli mutazionali negli isolati clinici di P. aeruginosa, i ricercatori hanno identificato batteri che presentavano già multiresistenza o che erano altamente inclini ad acquisire la multiresistenza in futuro. Questi risultati suggeriscono che l’analisi delle firme mutazionali potrebbe essere utilizzata come approccio diagnostico per identificare i ceppi batterici ad alto rischio prima che diventino intrattabili.
La capacità di prevedere se un ceppo batterico è destinato a sviluppare la multiresistenza rappresenta un passo fondamentale. Ciò, infatti, consente ai medici di intervenire tempestivamente con dei trattamenti mirati, invece di aspettare il fallimento delle terapie antibiotiche standard.
Prevenire la resistenza multipla ai farmaci: una nuova strategia
Uno degli aspetti più promettenti dello studio è la scoperta che le terapie combinate possono prevenire l’acquisizione della multiresistenza nei ceppi ipermutatori di P. aeruginosa. I ricercatori hanno scoperto che la combinazione di antibiotici con meccanismi d’azione diversi—come l’associazione di un antibiotico tradizionale con un peptide antimicrobico—ha significativamente ridotto la capacità del batterio di sviluppare resistenza.
Questo contrasta con il tradizionale ciclo di antibiotici, che spesso prevede l’alternanza tra due farmaci. Lo studio ha invece evidenziato che tali strategie di ciclo potrebbero effettivamente promuovere la resistenza anziché ridurla. Questa scoperta è particolarmente rilevante per i pazienti con fibrosi cistica, in cui le infezioni da P. aeruginosa vengono comunemente trattate con antibiotici inalatori a rotazione.
Combinando strategicamente farmaci che richiedono diversi meccanismi di resistenza, è possibile ridurre la probabilità che i batteri sviluppino resistenza multipla.
Oltre la fibrosi cistica: implicazioni più ampie
Sebbene lo studio si sia concentrato principalmente su P. aeruginosa nei pazienti con fibrosi cistica, le sue implicazioni si estendono ben oltre questa specifica patologia. L’analisi di genomi batterici disponibili pubblicamente ha rivelato che ceppi privi di MMR sono presenti anche in altri contesti infettivi, tra cui le infezioni del tratto respiratorio, le infezioni del tratto urinario e le infezioni intra-addominali. Questo suggerisce che la carenza di MMR potrebbe giocare un ruolo più ampio nella resistenza agli antibiotici in diverse infezioni batteriche.
Inoltre, i risultati sollevano preoccupazioni su come le strategie di trattamento antibiotico standard possano involontariamente selezionare ceppi ipermutatori. Poiché la resistenza agli antibiotici continua ad aumentare a livello globale, comprendere i meccanismi genetici ed evolutivi alla base dell’acquisizione della multiresistenza è fondamentale per sviluppare delle contromisure efficaci.
Un passo verso la medicina di precisione
I risultati dello studio si allineano a una tendenza crescente nella medicina: sfruttare i dati genomici per approcci terapeutici personalizzati. Se implementata nella pratica clinica, l’analisi delle firme mutazionali potrebbe aiutare i medici a identificare precocemente le infezioni inclini alla multiresistenza, consentendo di avere dei piani terapeutici su misura per minimizzare il suo sviluppo.
Attualmente, il sequenziamento dell’intero genoma sta diventando più accessibile per l’uso clinico, aprendo la strada all’analisi in tempo reale delle firme mutazionali negli ospedali. Integrando questa tecnologia con la diagnostica dei pazienti, i clinici potrebbero passare dal trattamento antibiotico reattivo a un approccio proattivo, basato sulla precisione.
Guardando al futuro
Mentre la lotta contro l’antibiotico-resistenza si intensifica, i risultati di questo studio evidenziano l’importanza della “sorveglianza genetica” nelle infezioni batteriche. La ricerca futura dovrebbe potrebbe concentrarsi sull’espansione dell’analisi delle firme mutazionali ad altri patogeni batterici ed esplorare il suo potenziale negli approcci diagnostici in tempo reale.
Questa ricerca rappresenta un passo nella giusta direzione. Identificare precocemente i ceppi batterici ad alto rischio consente, infatti, di affinare le strategie terapeutiche per contrastare la resistenza prima che diventi un problema insormontabile.
Di fronte a una crisi in evoluzione della resistenza agli antibiotici, l’innovazione degli strumenti diagnostici e degli approcci terapeutici è più cruciale che mai. L’analisi delle firme mutazionali offre una strada promettente verso la medicina di precisione, garantendo che gli antibiotici salvavita rimangano efficaci anche per le generazioni future.




